Support Vector Machines
O Support Vector Machines (SVM) é um algoritmo de aprendizado de máquina supervisionado baseado em kernels que é utilizado para problemas de classificação. Basicamente, o SVM encontra uma linha de separação, usualmente conhecida como hiperplano entre dados pertencentes de duas classes arbitrárias. Essa linha tenta maximizar a distância entre os pontos mais próximos em relação a cada uma das classes. Nesta versão, implementa somente uma versão específica do SVM linear. Por esse motivo, somente suporta problemas de classificação multi-classe (i.e., com mais de duas classes) caso seja realizada classificação um-contra-todos.
Conectores
| Entrada | Saída |
|---|---|
| Dados utilizados para treinar o modelo | Dados de saída e Modelo do algoritmo de classificação |
Tarefa
Nome da Tarefa
Aba Execução
| Parâmetro | Detalhe |
|---|---|
| Atributo com Features | Atributo(s) que será(ão) usado(s) para treinamento |
| Atributo com o rótulo | Atributo a ser classificado |
| Atributos com a predição (novo) | Atributo contendo a predição do modelo |
| Pesos | Peso do algoritmo em um Ensemble |
| Iterações máximas | Número máximo de iterações (>=0) |
| Aplicar padronização | Indica se é para padronizar os atributos antes de treinar o modelo |
| Limiar para a classificação binária | Ajustar a probabilidade de previsão de cada classe |
| Tolerância para a convergência | Tolerância de convergência para algoritmos iterativos (>=0.0) |
| Atributo com pesos | Atributos passados ao modelo que terão um peso diferenciado |
| Métrica para validação cruzada | Define a métrica utilizada dentro da validação cruzada (se aplicável) para avaliar o modelo de classificação dentro das k partições |
| Atributo com o número da partição (fold) | Define o atributo a ter o número da partição para realizar uma validação cruzada (se aplicável) |
| Usar classificação um-contra-todos (one-vs-rest) | Se selecionado, o algoritmo realizará classificação um-contra-todos ao invés de classificação tradicional (neste caso, binária) |
Exemplo de Utilização
Objetivo: Utilizar o modelo do Support Vector Machines (SVM) para classificar se uma pessoa possui ou não a doença diabetes
Base de Dados: Pima Indians Diabetes
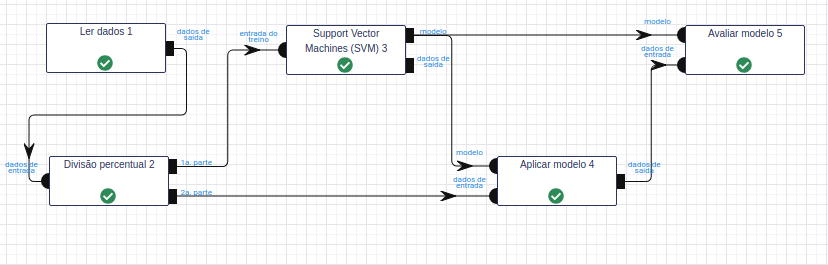
Leia a base de dados por meio da operação Ler dados.
Utilize a operação Divisão percentual para dividir a base de dados em treino e teste. No parâmetro Percentual, calibre-o utilizando 50% dos dados para treinar (1.ª parte) e 50% para testar (2.ª parte).
Na operação Support Vector Machines (SVM), selecione “Age”, “BMI”, “BloodPressure”, “DiabetesPedigreeFunction”, “Glucose”, “Insulin”, “Pregnancies” e “SkinThickness” no campo Atributos com features. Selecione “class” no campo Atributo com o rótulo e preencha “resultado” no campo Atributo com a predição (novo). Deixe os demais parâmetros inalterados.
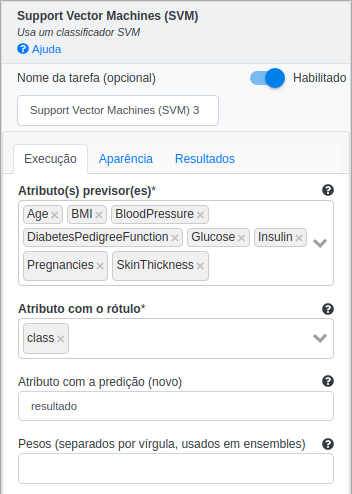
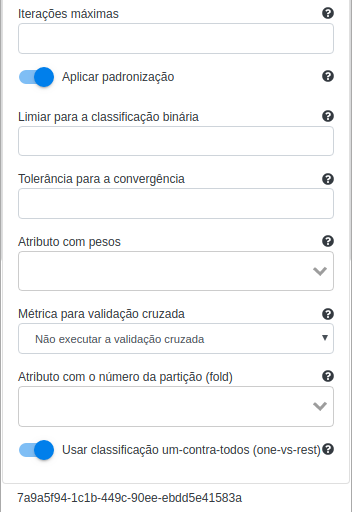
Na operação Aplicar Modelo, selecione “Age”, “BMI”, “BloodPressure”, “DiabetesPedigreeFunction”, “Glucose”, “Insulin”, “Pregnancies” e “SkinThickness” no campo Atributos com features e preencha “resultado” no campo Nome do novo atributo (herdado do modelo).
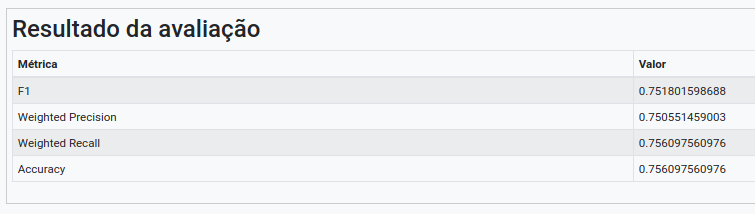
Na operação Avaliar Modelo, selecione “resultado” no campo Atributo usado para predição. Selecione “class” no campo Atributo usado como label e a métrica “F1” como Métrica para avaliação.
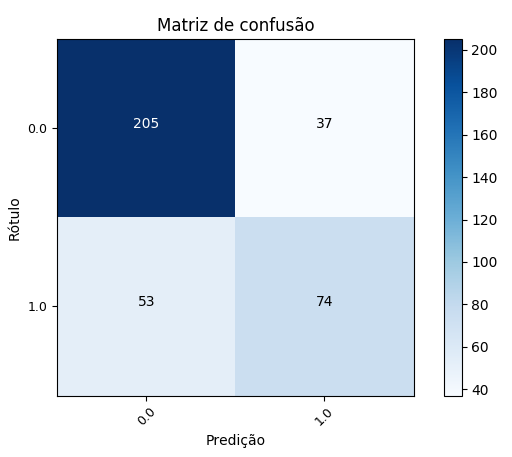
Execute o fluxo e visualize o resultado, i.e., a matriz de confusão gerada para as predições do modelo de árvore de decisão e, consequentemente, a tabela representando as métricas de classificação (derivadas da matriz de confusão).
Dúvidas e/ou sugestões envie um e-mail para suporte@lemonade.org.br
