Support Vector Machines
O Support Vector Machines (SVM) é um algoritmo de aprendizado de máquina supervisionado baseado em kernels que é utilizado para problemas de classificação. Basicamente, o SVM encontra uma linha de separação, usualmente conhecida como hiperplano entre dados pertencentes de duas classes arbitrárias. Essa linha tenta maximizar a distância entre os pontos mais próximos em relação a cada uma das classes. Nesta versão, implementa somente uma versão específica do SVM linear. Por esse motivo, somente suporta problemas de classificação multi-classe (i.e., com mais de duas classes) caso seja realizada classificação um-contra-todos.
Conectores
| Entrada | Saída |
|---|---|
| Dados utilizados para treinar o modelo | Dados de saída e Modelo do algoritmo de classificação |
Tarefa
Nome da Tarefa
Aba Execução
| Parâmetro | Detalhe |
|---|---|
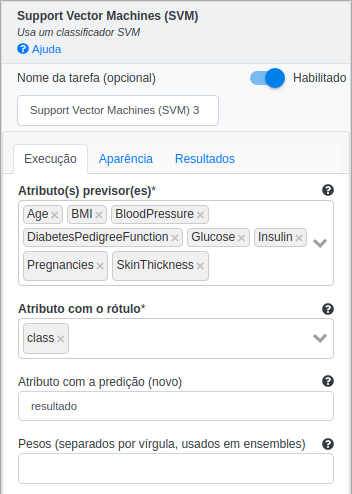
| Atributo com Features | Atributo(s) que será(ão) usado(s) para treinamento |
| Atributo com o rótulo | Atributo a ser classificado |
| Atributos com a predição (novo) | Atributo contendo a predição do modelo |
| Pesos | Peso do algoritmo em um Ensemble |
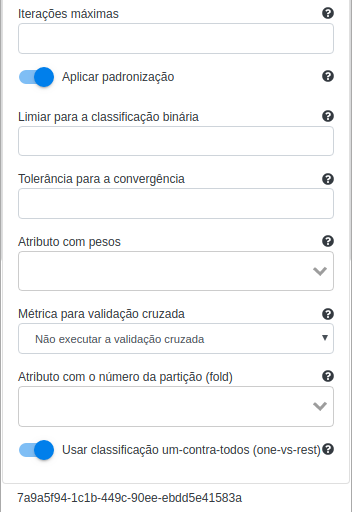
| Iterações máximas | Número máximo de iterações (>=0) |
| Aplicar padronização | Indica se é para padronizar os atributos antes de treinar o modelo |
| Limiar para a classificação binária | Ajustar a probabilidade de previsão de cada classe |
| Tolerância para a convergência | Tolerância de convergência para algoritmos iterativos (>=0.0) |
| Atributo com pesos | Atributos passados ao modelo que terão um peso diferenciado |
| Métrica para validação cruzada | Define a métrica utilizada dentro da validação cruzada (se aplicável) para avaliar o modelo de classificação dentro das k partições |
| Atributo com o número da partição (fold) | Define o atributo a ter o número da partição para realizar uma validação cruzada (se aplicável) |
| Usar classificação um-contra-todos (one-vs-rest) | Se selecionado, o algoritmo realizará classificação um-contra-todos ao invés de classificação tradicional (neste caso, binária) |
[Aba Aparência][1]
[Aba Resultados][2]
Exemplo de Utilização
Objetivo: Utilizar o modelo do Support Vector Machines (SVM) para classificar se uma pessoa possui ou não a doença diabetes
Base de Dados: [Pima Indians Diabetes][3]
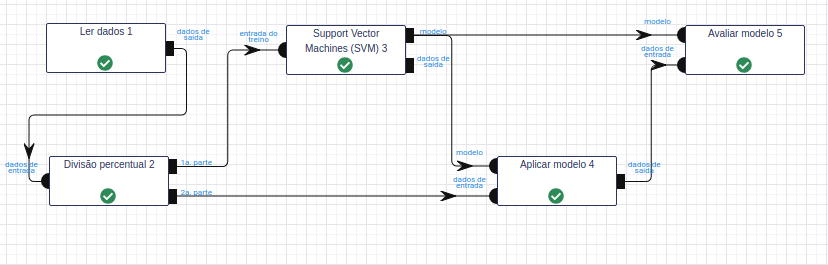
Leia a base de dados por meio da operação [Ler dados][4].
Utilize a operação [Divisão percentual][5] para dividir a base de dados em treino e teste. No parâmetro Percentual, calibre-o utilizando 50% dos dados para treinar (1.ª parte) e 50% para testar (2.ª parte).
Na operação Support Vector Machines (SVM), selecione “Age”, “BMI”, “BloodPressure”, “DiabetesPedigreeFunction”, “Glucose”, “Insulin”, “Pregnancies” e “SkinThickness” no campo Atributos com features. Selecione “class” no campo Atributo com o rótulo e preencha “resultado” no campo Atributo com a predição (novo). Deixe os demais parâmetros inalterados.
Na operação [Aplicar Modelo][6], selecione “Age”, “BMI”, “BloodPressure”, “DiabetesPedigreeFunction”, “Glucose”, “Insulin”, “Pregnancies” e “SkinThickness” no campo Atributos com features e preencha “resultado” no campo Nome do novo atributo (herdado do modelo).
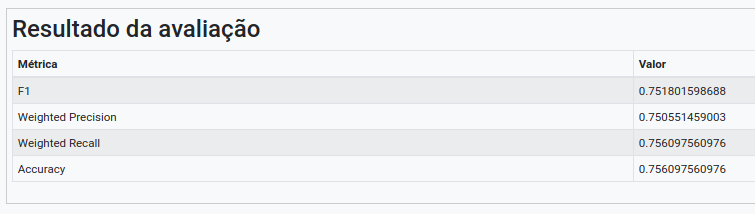
Na operação [Avaliar Modelo][7], selecione “resultado” no campo Atributo usado para predição. Selecione “class” no campo Atributo usado como label e a métrica “F1” como Métrica para avaliação.
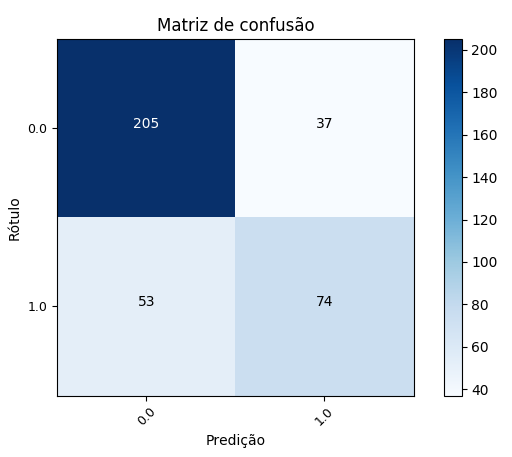
Execute o fluxo e visualize o resultado, i.e., a matriz de confusão gerada para as predições do modelo de árvore de decisão e, consequentemente, a tabela representando as métricas de classificação (derivadas da matriz de confusão).
Dúvidas e/ou sugestões envie um e-mail para suporte@lemonade.org.br
